使用LTR装配指数(LAI)评估基因组组装质量
使用LTR装配指数(LAI)评估基因组组装质量
基因组组装完成之后,就需要对最后的质量进行评估。我们希望得到的 contig 文件中,每个 contig 都能足够的长,能够有一个完整的基因结构,归纳一下就是3C原则:
- 连续性(Contiguity): 得到的contig要足够的长
- 正确性(Correctness): 组装的contig错误率要低
- 完整性(Completeness):尽可能包含整个原始序列
但是这三条原则其实是相互矛盾的,连续性越高,就意味着要处理更多的模糊节点,会导致整体错误率上升,为了保证完全的正确,那么就会导致contig非常的零碎。此外,这三条原则也比较定性,我们需要更加定量的数值衡量,目前比较常用的标准是 N50 和 BUSCO/CEGMA。
那有没有其他方法可以用来评估基因组装的质量呢,文章 “Assessing genome assembly quality using the LTR Assembly Index (LAI)“ 提出了用长末端重复序列来评估基因组完整度,因为LTR比较难以组装,于是就用作评估结果的一个参数了。
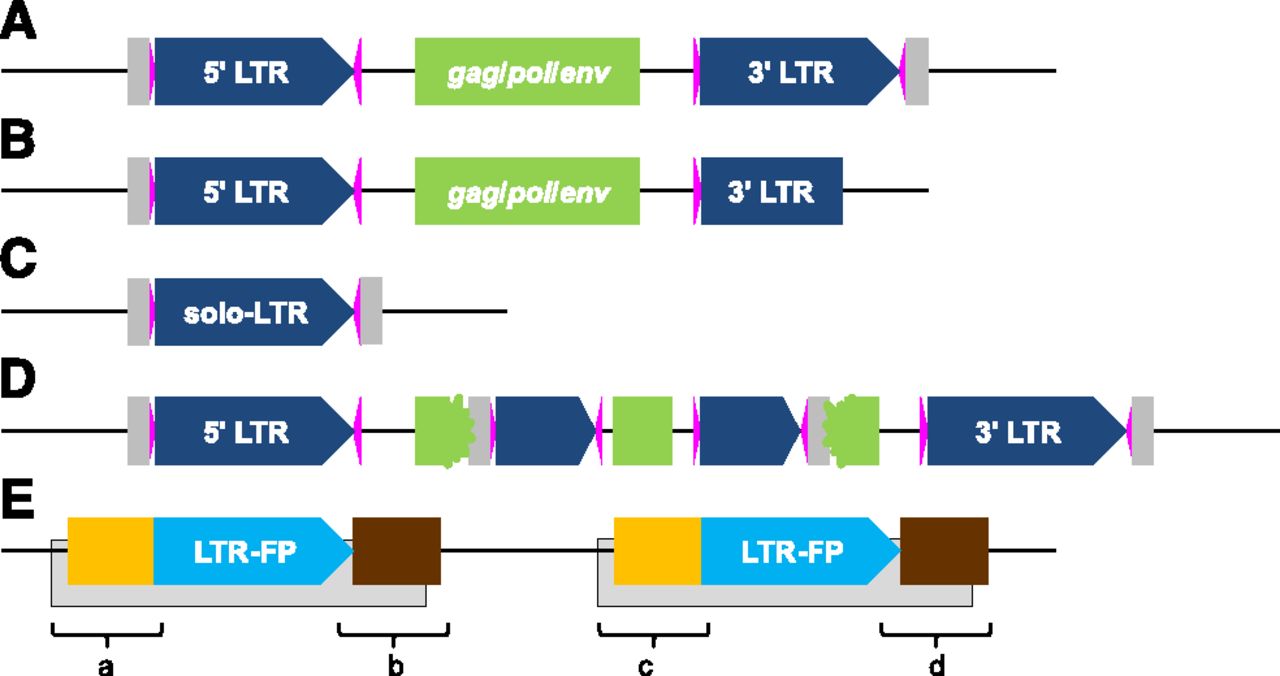
LTR(long terminal repeat)即长末端重复序列,其长度从 100bp 到 5kb 不等。是存在于LTR反转录转座子(LTRs)两侧翼的长末端重复序列。LTR反转录转座子(LTR-RTs)和非LTR反转录转座子(non-LTR)都是真核生物中一类可移动因子,因其转座需经由RNA介导的反转录过程而得名。LTR反转录转座子一高拷贝在生物界广泛分布,可以通过纵向和横向分别在世代之间和不种间进行传递,同一家族的反转录转座子具有高度的异质性。
LAI 指数就是完整 LTR 反转座子序列占总 LTR 序列长度的比值。
计算 LAI 的软件主要是 LTR_retriever 软件, 该软件可用于识别长末端重复反转录转座子。LTR_retriever 不是一个独立的工具,他的主要作用就是整合 LTRharvest, LTR_FINDER 和/或 MGEScan_LTR 的结果,过滤其中的假阳性 LTR-RT,得到高质量的 LTR-RT 库。
1. 软件安装
1.1 快速安装
参考官方安装说明,可以使用 conde 快速安装相关依赖及软件。
1 | conda create -n LTR_retriever |
1.2 标准安装
LTR_retriever 依赖于其他软件:
1 | 1. makeblastdb, blastn, and blastx in the BLAST+ package (ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/), |
下载安装 LTR_retriever 软件及依赖软件后,编辑 LTR_retriever/paths 文件,添加各依赖文件的路径:
1 | BLAST+=/your_path_to/BLAST+2.2.30/bin/ |
2. LTR_retriever 的使用
2.1 找到基因组 LTR
第一步首先需要找到基因组总的 LTR,使用 LTR_FINDER 程序进行查找:
1 | $ ltr_finder -D 20000 -d 1000 -L 700 -l 100 -p 20 -C -M 0.9 assembled.fa >out.finder.scn |
2.2 生成非冗余LTR-RT文库
第二步运行LTR_retriever根据LTR_FINDER的输出识别LTR-RT,生成非冗余LTR-RT文库,可用于基因组注释.
1 | $ LTR_retriever -threads 4 -genome assembled.fa -infinder out.finder.scn |
2.3 计算 LAI
依据第二三步的结果进行 LAI 的计算:
1 | LAI -t 10 -genome assembled.fa -intact out.fa.pass.list -all out.txt |
3. 项目分析
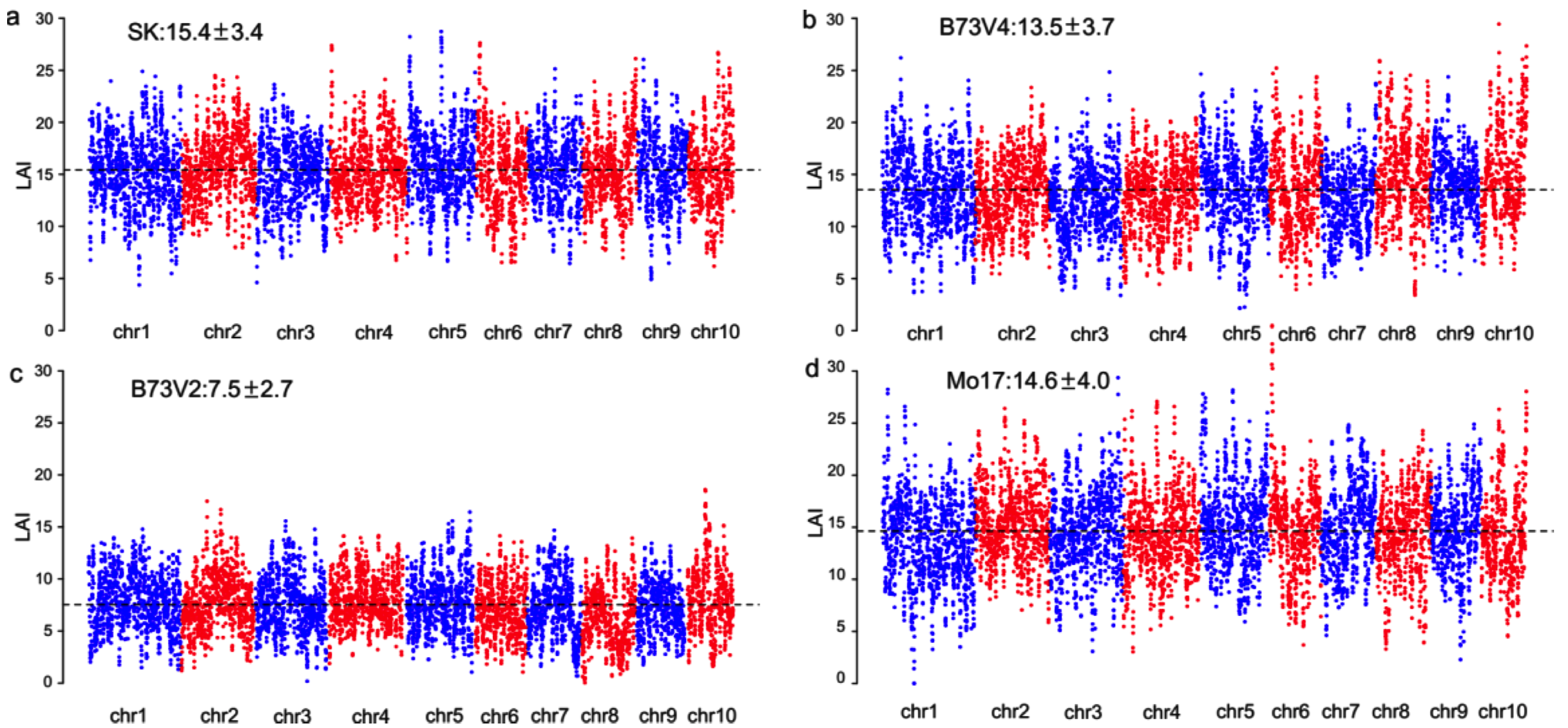
通过三代基因组组装,我们组装出了自己参考物种的基因组,maize_GF.S.fa, 然后使用 LAI 评估组装质量。计算参数参考严建兵老师 NG 文章(热带玉米基因组)的程序参数计算。
经过计算,得到 LAI 为 ~
与玉米参考基因组比对,LAI 装配指数达到中等水平,基因组可用。
参考资料
- LAI:评估基因组质量一个标准 https://www.jianshu.com/p/7d794d22e0a0
- LTR_retriever: 一个更加准的LTR整合分析工具 https://www.jianshu.com/p/f962d5c40fdf
- Github LTR_retriever: https://github.com/oushujun/LTR_retriever
- 严建兵老师热带玉米组装: https://www.nature.com/articles/s41588-019-0427-6
- LAI 参考文献: doi:10.1093/nar/gky730
- LTR_retriever 参考文献: doi:10.1104/pp.17.01310


