【生信分析】不同过滤软件的比较
1. 起缘
小刘哥在分析拿到的 Small RNA 数据时发现不同的过滤软件对数据的过滤结果并不一样。由此我们想找到到底什么样的参数、哪款软件能满足我们 Small RNA 数据过滤的基本需求。抱着这样的目的我们做了下面的测试:
使用的数据是我们一个真实的玉米根系数据,使用 NextSeq500/NextSeq500AR SE50 进行的 Small RNA 测序。
2. 原始数据测序质量
我们首先使用 fastqc 对数据的测序质量来个基本的了解。
1 | fastqc sample.fq.gz |
生成网页报告如下:
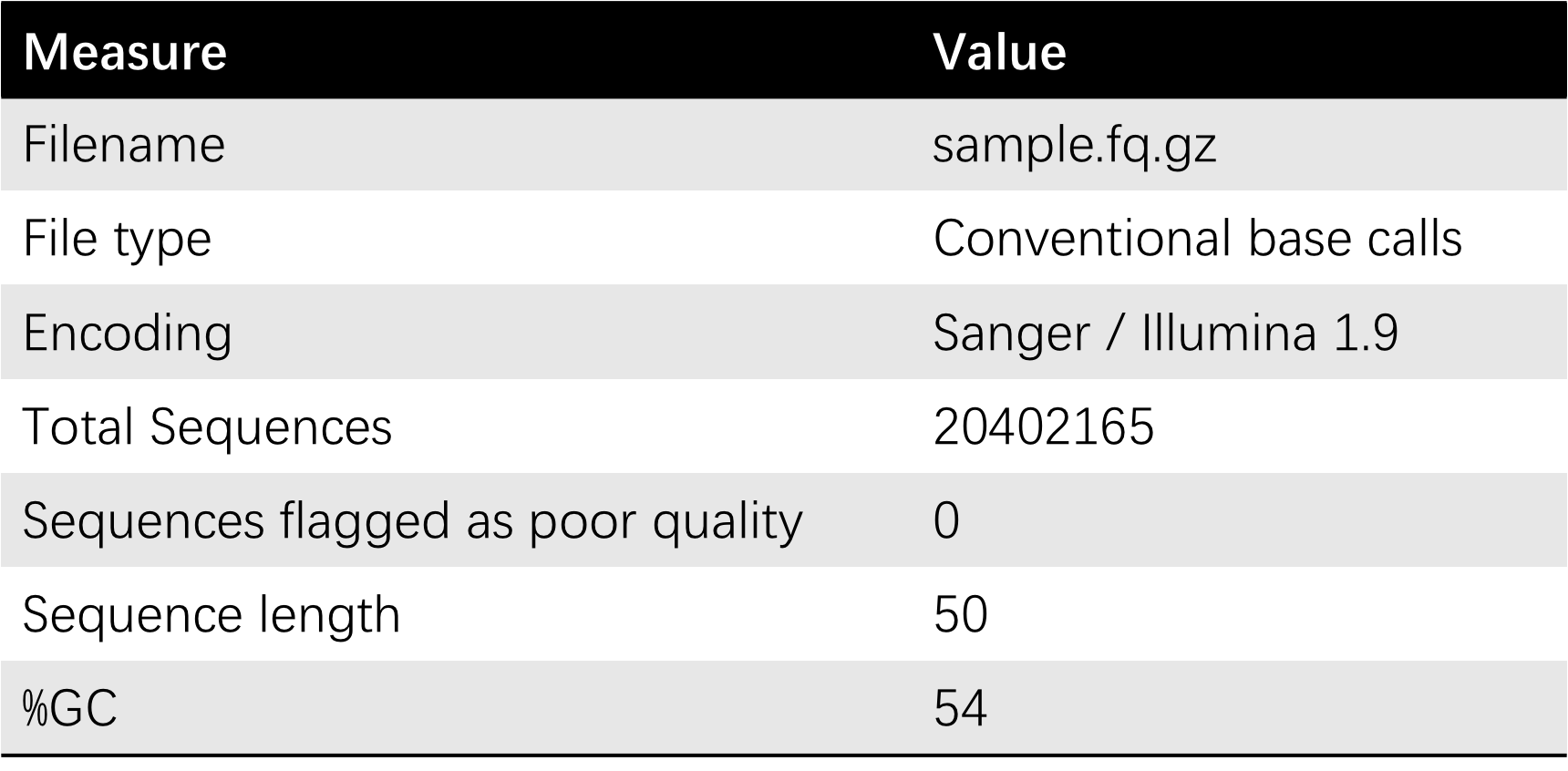
数据统计发现共有原始数据 20,402,165 条,既 20.4M 原始reads。原始数据的 GC含量为 54%,测序长度为 50bp,数据质量格式为 Sanger / Illumina 1.9 格式 既 phred33 格式。
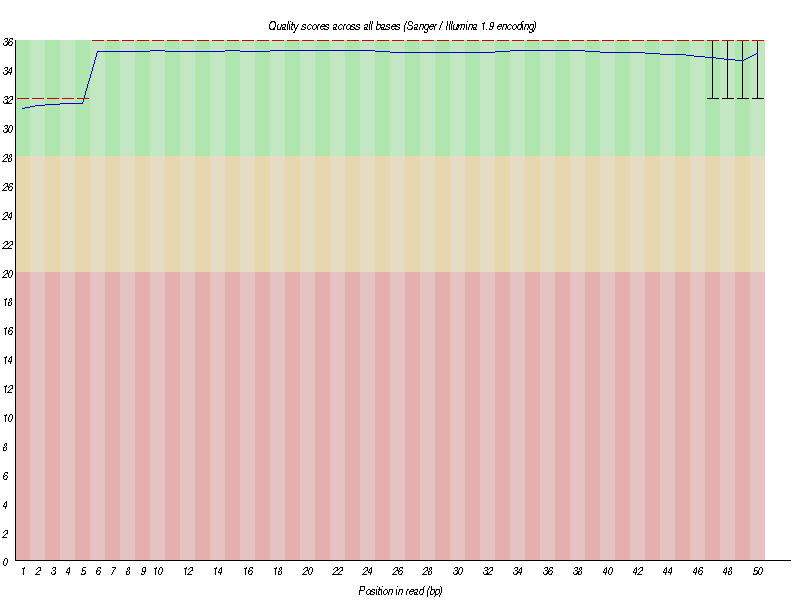
数据质量如下图:
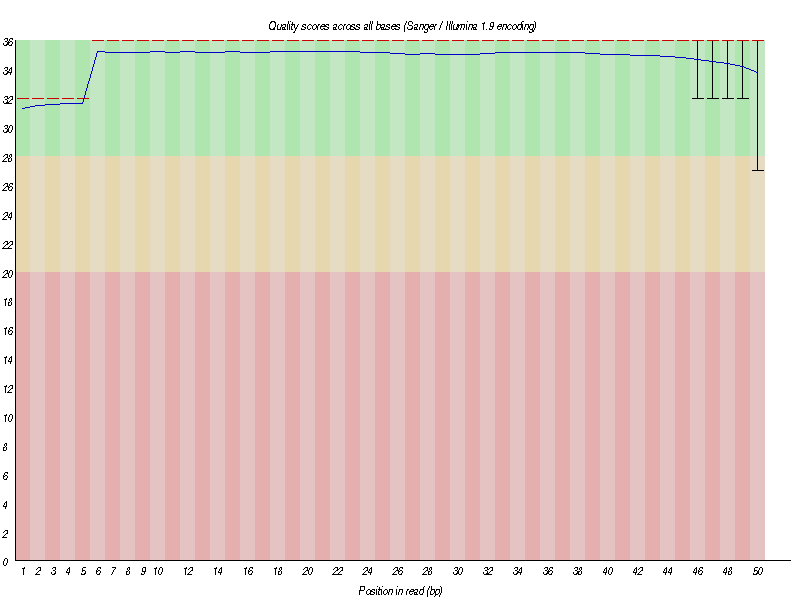
整体测序质量高,绝大部分碱基质量分数在 30 分以上,既测序的准确率在 99.9% 以上。
没有测出的碱基(N碱基)几乎为 0:
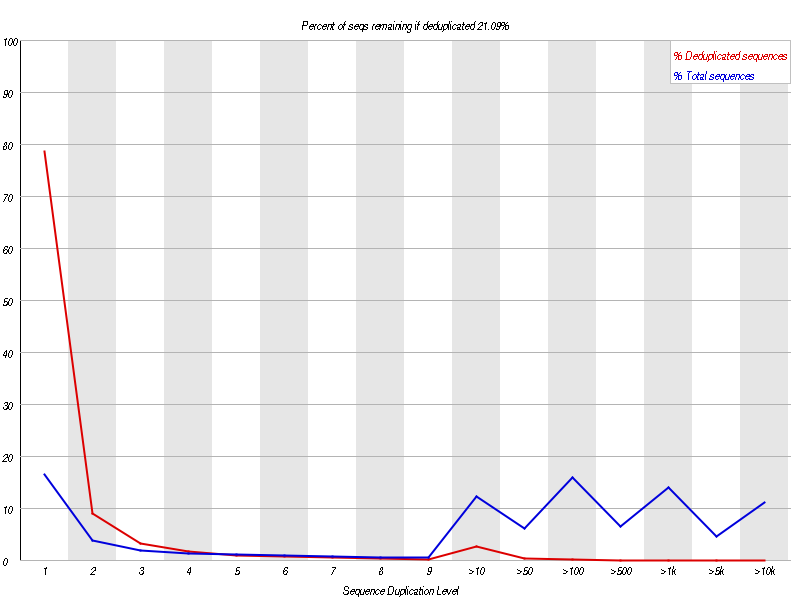
重复序列占总序列的 21.09% ,这是因为 Small RNA 测序文库片段较短,因此重复性较高。
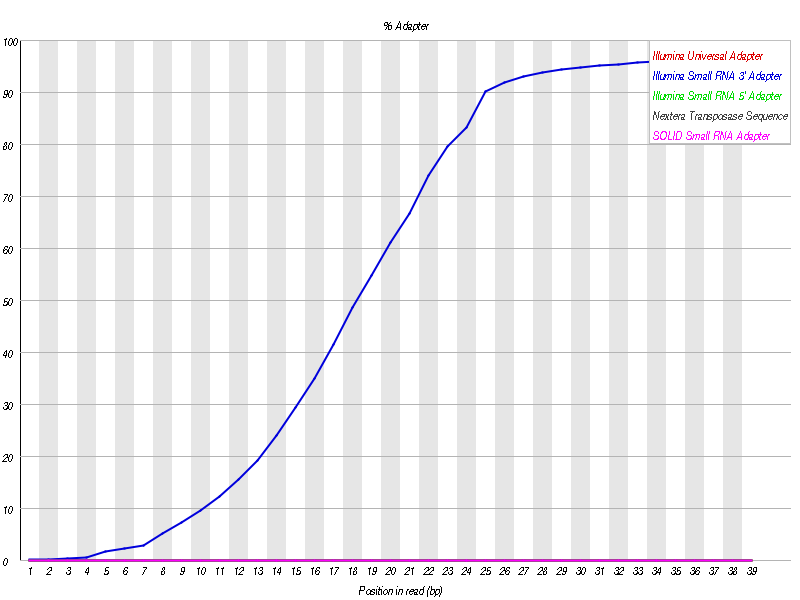
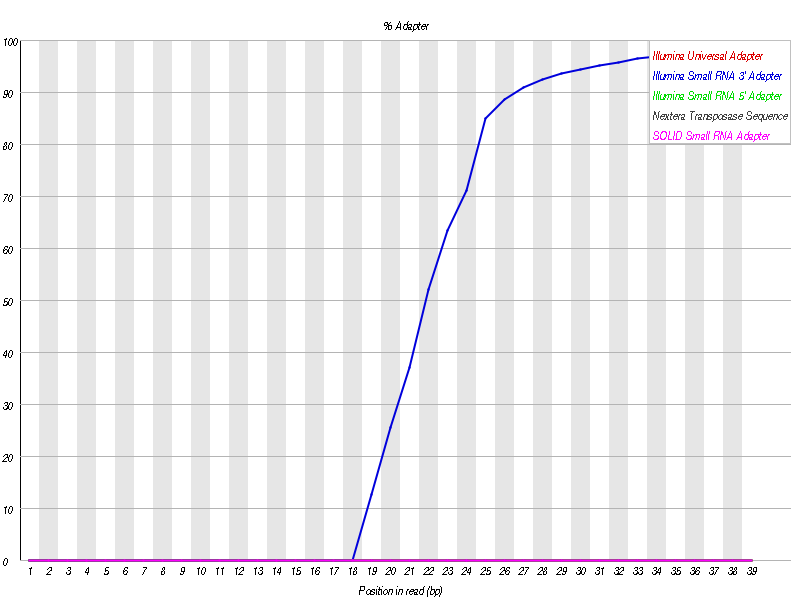
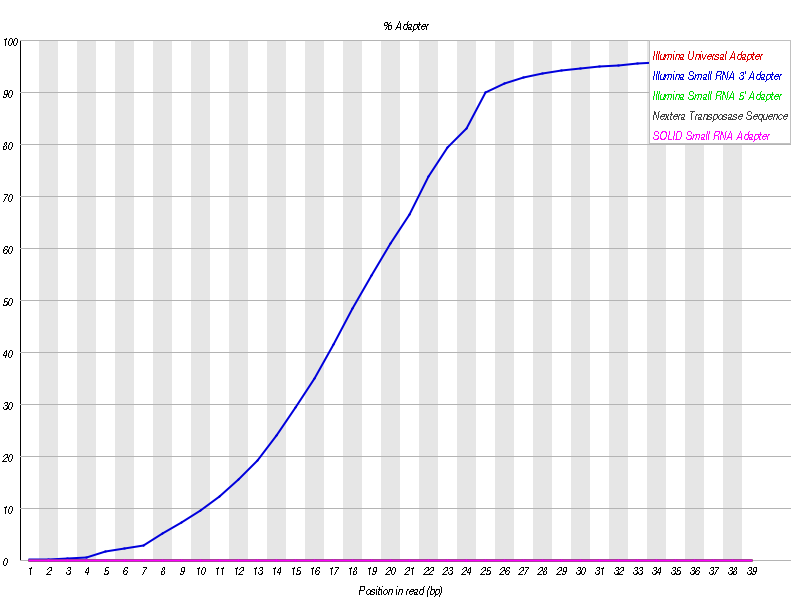
我们发现测序数据中主要包含了 Illumina Small RNA 3’ Adapter:
接下来,我们就使用不用的软件对这个数据进行低质量数据过滤,并比较其过滤结果。
3. 数据过滤标准
考虑到经常分析的 Small RNA 的有效长度基本我为 18 - 35 nt, 我们统一使用以下的过滤标准:
- 去除低质量 reads( sQ <= 20 的碱基数占整个 Reads 的 40% 以上)
- 去除 N 碱基比例大于 5 的 Reads
- 去除含有 5‘ 接头污染的 reads
- 去除没有 3‘ 接头的序列
- 去除插入片段缺失的 reads
- 去除 3‘ 接头
- 去除 Ploy/T/C/G 的序列
- 去除过长以及过短的 Reads (18 <= length <= 35)
4. 使用 SOAPnuke 进行过滤
SOAPnuke 是华大的老牌软件了,有专门过滤 small RNA 序列的模块,下载地址:https://github.com/BGI-flexlab/SOAPnuke 安装过程参照其说明文档。
具体参数使用以下命令查看:
1 | SOAPnuke filtersRNA -h |
{ % asset_img 72829788.png SOAPnuke 命令 % }
使用以下命令进行过滤:
1 | SOAPnuke filtersRNA --fq1 sample.fq.gz --cleanFq1 Soap.Clean.fq.gz --trimFq1 trim.sample.fq.gz --outDir ./ --outFileType fastq --log SOAPnukeFilter.log --adapter1 GTTCAGAGTTCTACAGTCCGACGATC --adapter2 TGGAATTCTCGGGTGCCAAGG --seqType 1 --ada_trim --adaRCtg 6 --adaRAr 0.8 --adaRMa 5 --adaREr 0.4 --lowQual 20 --qualRate 0.4 --nRate 0.05 --mean 20 --polyA 0.4 --polyX 10 --index --thread 8 --qualSys 2 --outQualSys 2 --maxReadLen 35 --minReadLen 18 |
DELL R730xd 的二手服务器主机,E5-2678 v3 处理器(24核,48线程),我使用了 8 个线程,花费 1分 51秒 完成了过滤,同时生成了多个统计文件:
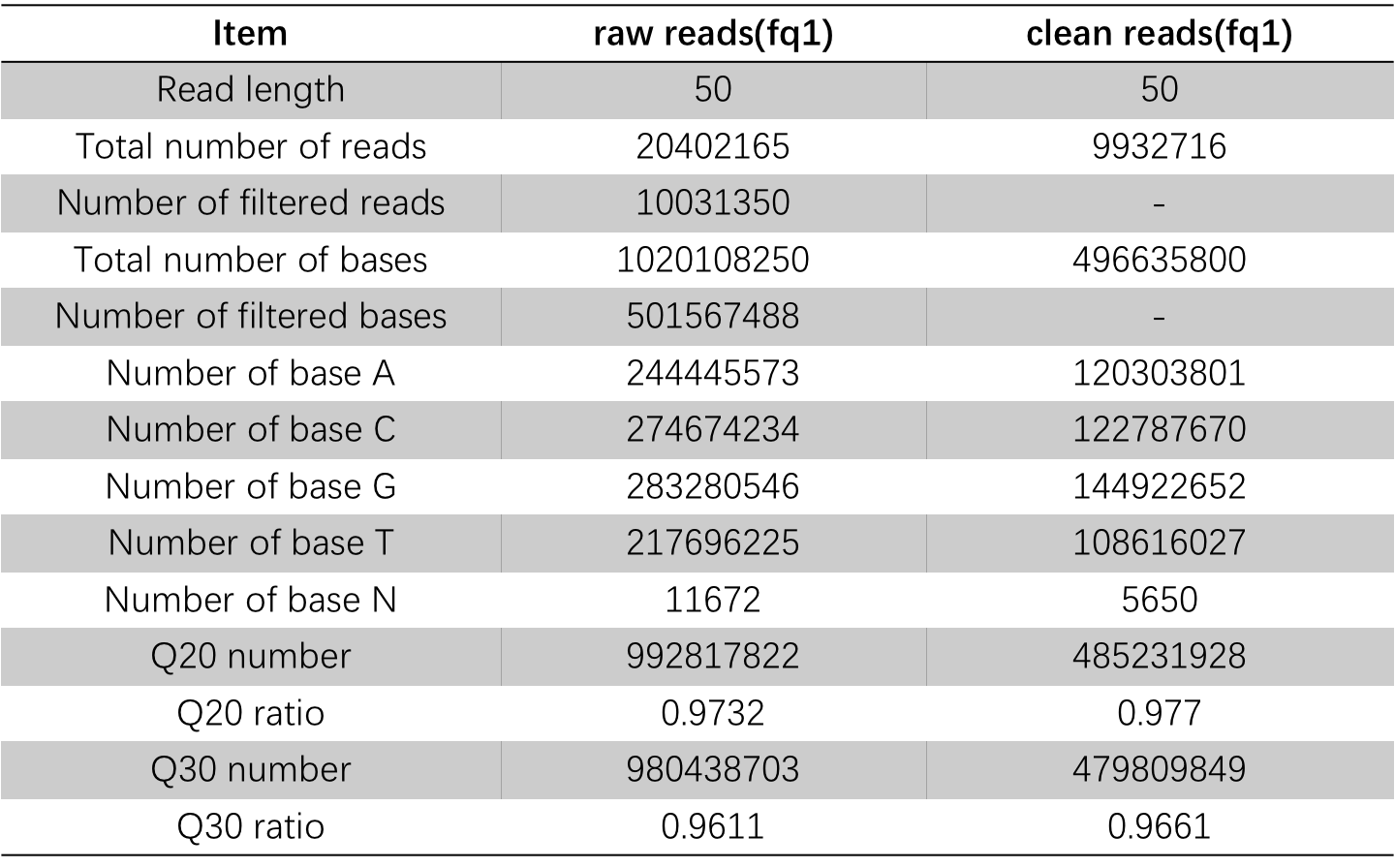
过滤完成后还剩余 9.93 M 的 Reads,过滤掉一半多。
我们还是使用 fastqc 软件再直观的查看一下过滤后的 Clean Reads 数据质量。
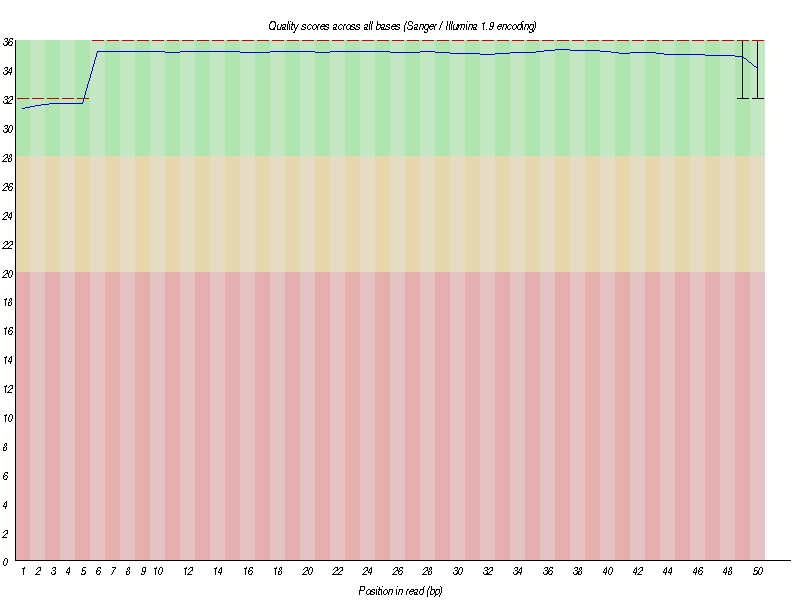
基本将低质量数据全部过滤掉了,遗憾的地方是,并没有将数据中的 接头序列剪切掉。
同时我们测试了不指定 Adapter 序列运行程序,发现程序对接头序列识别差的很,不得已,只有保留指定接头序列参数。
5. 使用 fastp 软件进行过滤
fastp 软件是海普洛斯公司研发团队开发的一个快速的过滤工具,下载地址:https://github.com/OpenGene/fastp
我使用 conda 安装了该软件:
1 | conda install fastp |
由于使用了指定的 Adapter 序列,导致运行结果严重失真,经测试发现 fastp 软件对单端数据默认只识别 RA3‘ 接头序列。
1 | fastp --in1 ../sample.fq.gz --out1 fastp.Clean.fq.gz --adapter_sequence TGGAATTCTCGGGTGCCAAGG --trim_poly_g --poly_g_min_len 10 --trim_poly_x --poly_x_min_len 10 --cut_window_size 5 --cut_mean_quality 20 --qualified_quality_phred 15 --unqualified_percent_limit 40 --n_base_limit 1 --length_required 18 --length_limit 35 --json fastp.Clean.json --html fastp.Clean.html --thread 8 |
用时 66 秒, 结果如下:
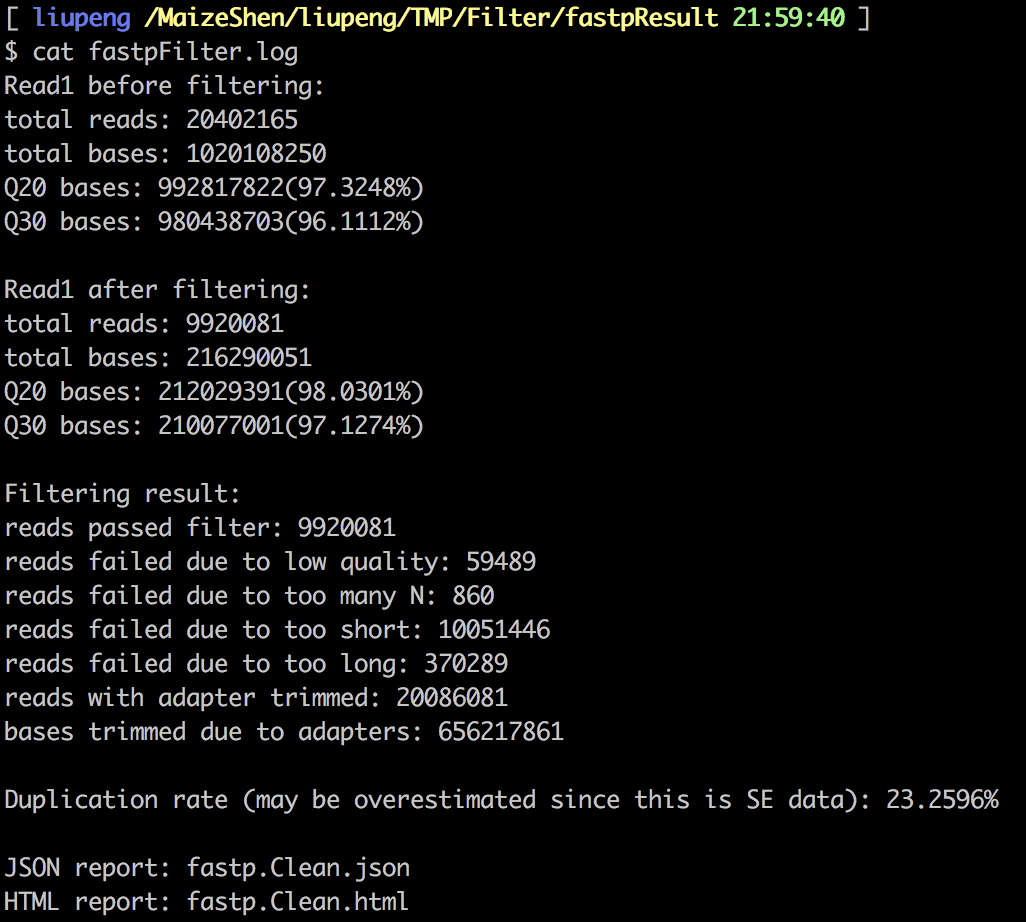
可以看到过滤完后还剩余 9.92M 的 Clean Reads,而且结果也是切好的数据。比 SOAPnuke 软件切掉的数据少一些。
然后我们使用不加 adapter 参数的命令重新过滤,运行时间:64 秒,同样的,没有识别到 Adpter 序列,把长度大于 35 bp的序列都过滤掉了,因此我们只有保留接头序列参数。
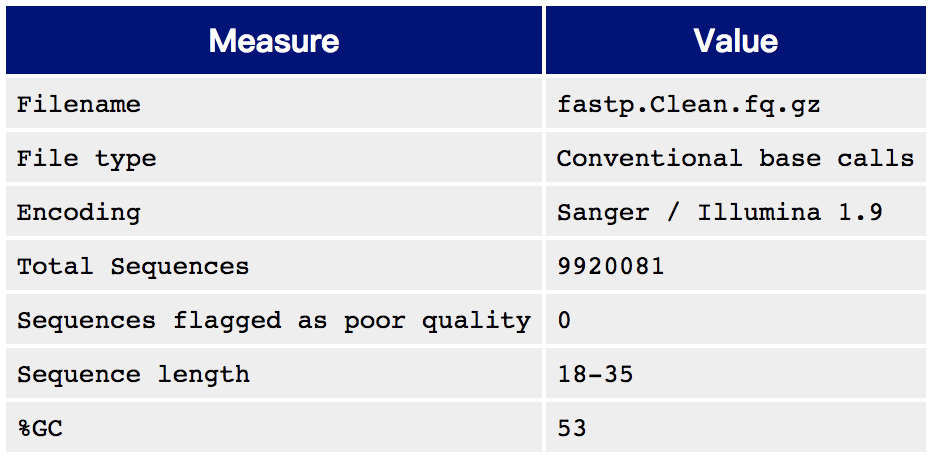
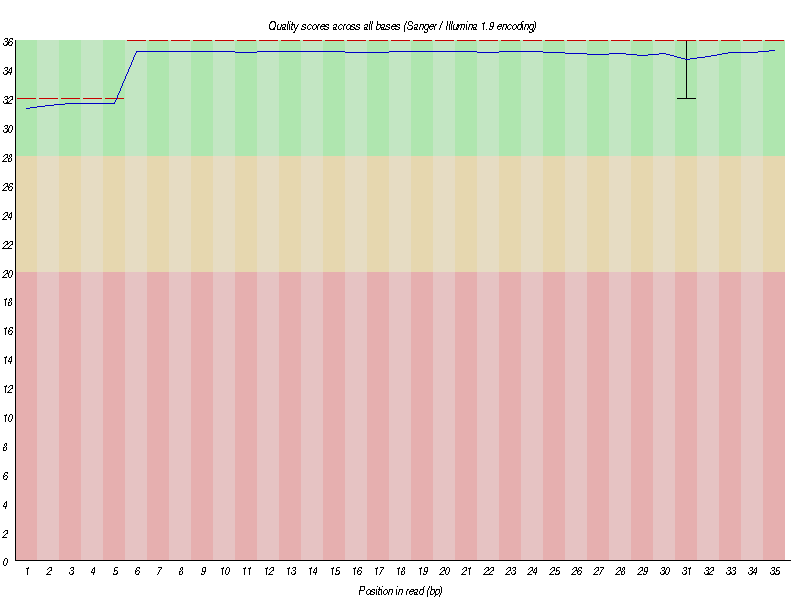
fastqc 对 过滤后的 clean Reads 进行检验:发现基本也将低质量数据过滤完全了,并且接头也去除干净了。
6. 使用 cutadapt 软件进行过滤
cutadapt 是一款经典的测序数据过滤软件了,基于 python 编写的。
使用 conda 进行安装:
1 | conda install cutadapt |
安装完成后使用如下命令进行过滤:
1 | cutadapt -a TGGAATTCTCGGGTGCCAAGG -j 8 -o cutadapt.Clean.fq.gz -m 18 -M 35 --max-n 2 --discard-untrimmed ../sample.fq.gz |
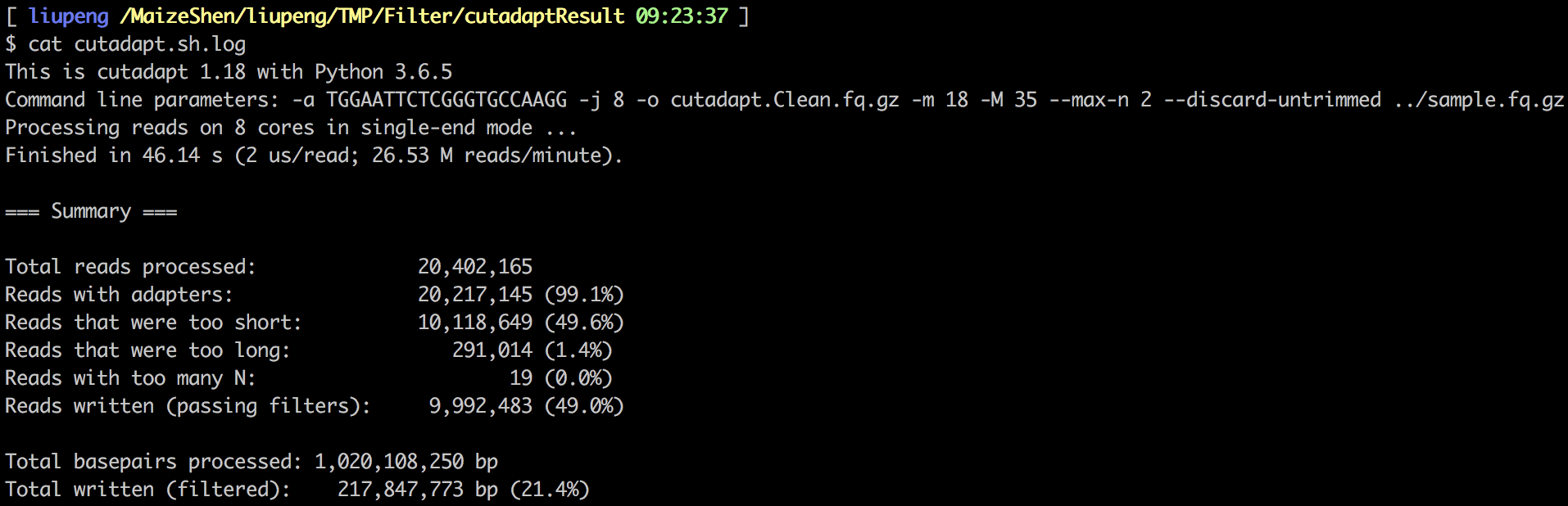
同样的 8 线程,用时46.14 秒,过滤完成后保留了 9.99M 的 Clean Reads。
同样的,我们使用 fastqc 对过滤后的数据进行查看:
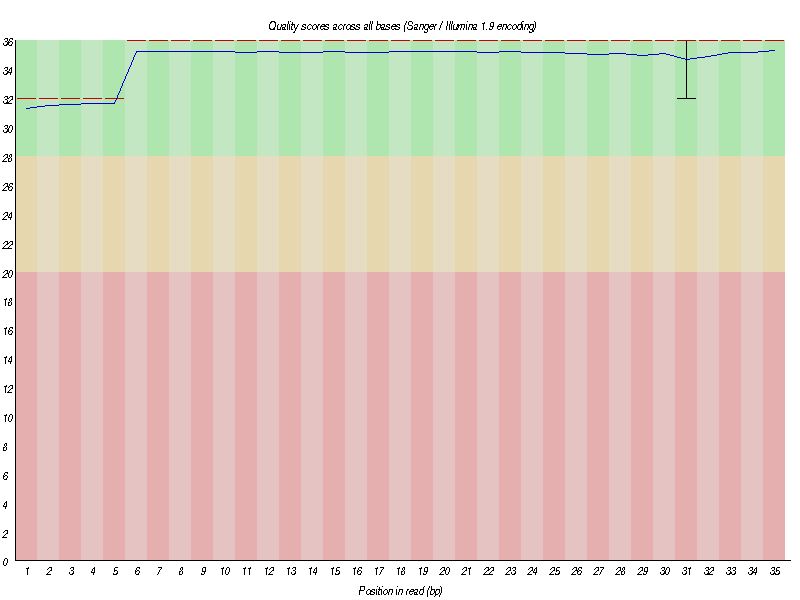
接头基本过滤干净了。
7. 使用 Trimmomatic 进行过滤
Trimmomatic 发表的文章至今已被引用了 6784 次,是一个广受欢迎的 Illumina 平台数据过滤工具。下载地址: http://www.usadellab.org/cms/index.php?page=trimmomatic
我们安装还是使用 conda :
1 | conda install trimmomatic |
我们使用一下命令进行过滤:
1 | trimmomatic SE -threads 8 -phred33 -trimlog trim.log -summary trim.stat ../sample.fq.gz Trimmomatic.Clean.fq.gz ILLUMINACLIP:/MaizeShen/liupeng/TMP/Filter/adapter.txt:2:30:10 LEADING:13 TRAILING:13 SLIDINGWINDOW:5:20 MINLEN:18 TOPHRED33 |
fastqc 检测结果如下:
其结果也无法将接头直接去掉。
综合考虑,我们还是使用 fastp 软件进行过滤。


