小刘哥学生信系列一数据质控
小刘哥学生信系列 — 从数据质控开始
前面两章我们配置了基本的运行环境,也安装了一些基本的运行环境,今天我们就主要与大家分享一下生信分析的第一步: 数据质控
对于我们经常接触的二代数据来说,不管是 DNA 还是转录组,下机数据一般都是给的我们 fastq 格式的原始数据。我们使用最经典的 fastqc 软件来查看数据的测序质量。
安装 fastqc 软件前,推荐大家首先安装 conda ,然后使用 conda 安装各类需要使用的软件。conda 因为可以解决好多软件之间的相关性,对我们动辄就需要编译源代码来安装的方法来说节省了很大的时间和精力。
推荐安装 Anacoda,也可以安装精简的 Miniconda,我选择安装 Miniconda,链接如下 :Miniconda — Conda,选择相应版本下载:

我们以 Linux 64位 Python3.6 版本为例,下载后得到一个 Miniconda3-latest-Linux-x86_64.sh 的文件,就以普通用户安装就可以了。
1 | sh Miniconda3-latest-Linux-x86_64.sh |
如下图:
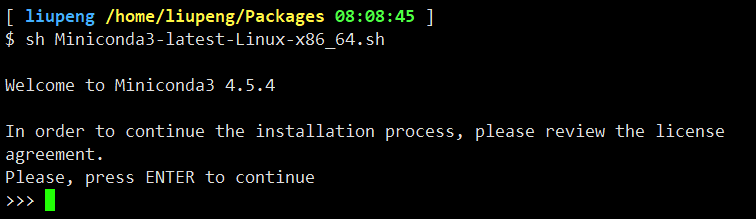
回车继续,然后输入 yes 同意安装协议,进行安装:
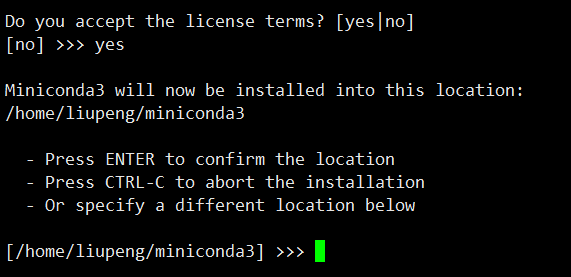
安装完成后,就可以在 /home/liupeng/miniconda3/bin 目录下找到 conda 命令。
加上绝对路径就可以使用了:
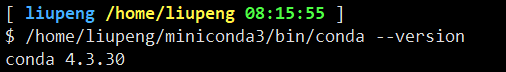
如果不喜欢加 绝对路径的话,就在环境变量 (PATH) 里加上一句就可以了:
1 | export PATH="/home/liupeng/miniconda3/bin:$PATH" |
安装好以后对 conda 进行简单配置就好了,因为咱们用的大多数的生信软件包都收录在了 bioconda 仓库中,所以我们在命令行中需要运行以下三条命令进行配置:
1 | conda config --add channels defaults |
配置完成后,到这里我们前期的准备工作就完成了,下面我们就开始安装生物信息的第一个软件: fastqc
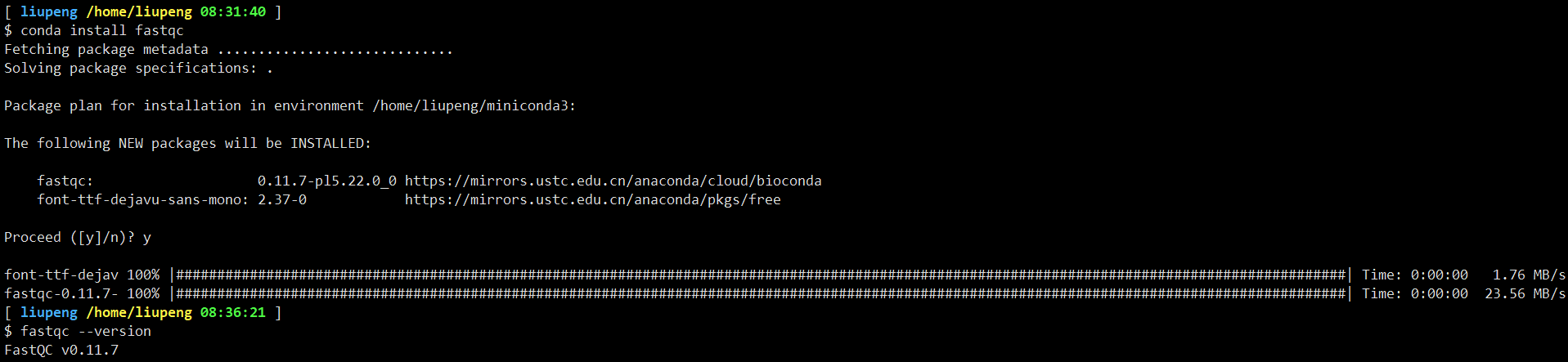
1 | conda install fastqc |
OK, 你没有看错,只需要这一条命令,就可以把 fastqc 软件安装上了。
然后使用 fastqc —version 检查程序是否安装无误:
安装安装无误后,我们就可以拿来分析我们的数据了,二代和三代的数据 fastqc 都可以进行质控,使用前我们先看看其可以使用哪些参数:
输入命令
1 | fastqc --help |
查看
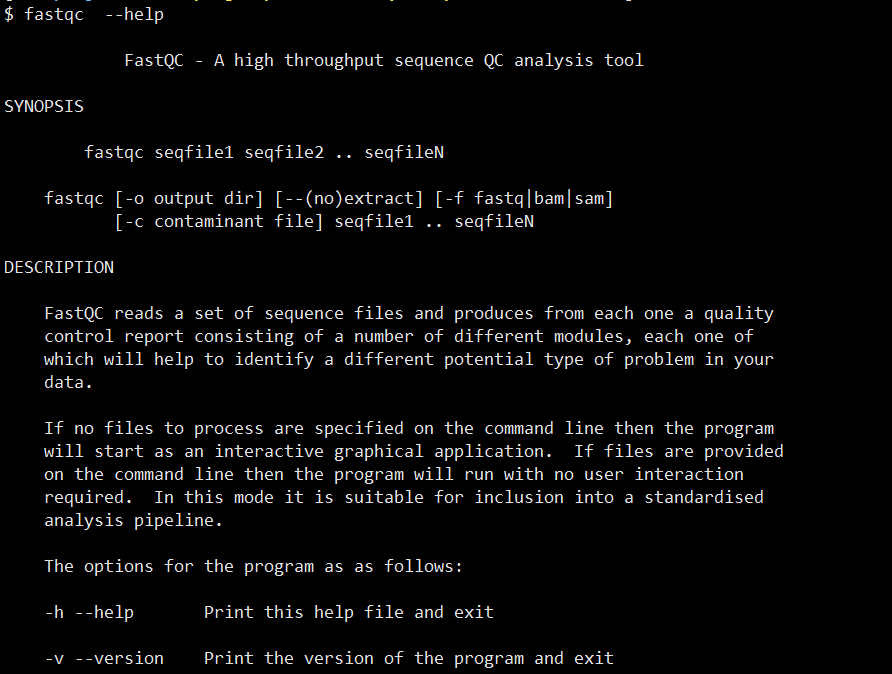
看明白每个参数有助于我们更好的使用软件。
我们使用一个比较少的参数先试一下:
1 | fastqc -o fastqcResult -t 4 sampe1.R1.fq.gz sampe1.R2.fq.gz |
如图:

运行完成后会在 fastqcResult 目录下生成四个文件:
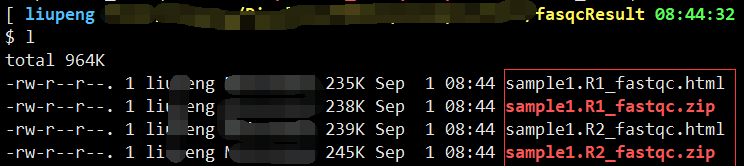
即每个数据文件生成一个 html 页面报告文件和一个 zip 压缩包
使用浏览器打开 html 报告文件后,我们可以看到如下图所示的质控结果:
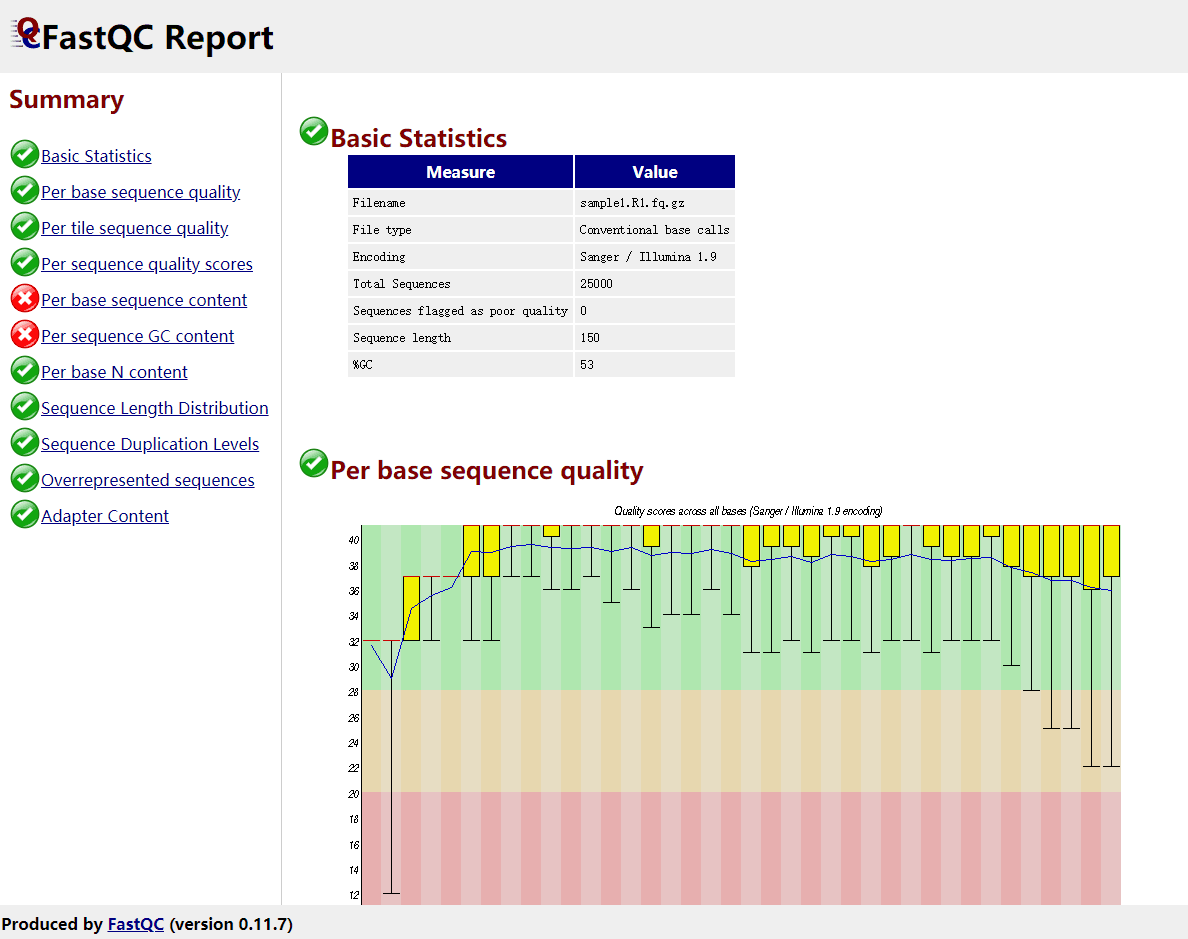
至于每一项所代表的意义,大家可以查看官方给出的帮助文档,解释的很详细了:
http://www.bioinformatics.babraham.ac.uk/projects/fastqc/Help/3%20Analysis%20Modules/
经过质控,对于不合格数据我们需要进一步使用过滤软件对低质量数据进行过滤,数据合格的话我们就可以进行后续的分析了。


